近日,我院杨朝勇教授课题组在单细胞谱系示踪领域取得新进展,相关成果以" LINEAGE:Label Free Identification of Endogenous Informative Single Cell Mitochondrial RNA Mutation for Lineage Analysis"为题,发表于Proceedings of the National Academy of Sciences(DOI: 10.1073/pnas.2119767119)。
谱系示踪技术是研究细胞发育、疾病发生以及癌细胞克隆进化的重要分析方法。随着单细胞转录组测序(scRNA-seq)技术和相关计算工具的发展,scRNA-seq数据已被应用于细胞谱系信息的挖掘。传统的谱系示踪技术和基于基因编辑的谱系分析都受限于复杂的实验操作,难以应用于临床样本;以全基因组上的RNA突变为内源性条形码的谱系分析方法又对测序覆盖度有高要求;相比之下,高频、可传代的线粒体基因组RNA突变是内源性条形码的较优选择,然而已有方法需要同批样本的群体染色质可及性测序(bulk ATAC-seq)或者全基因组测序数据进行谱系信息提取,同样限制了其应用。
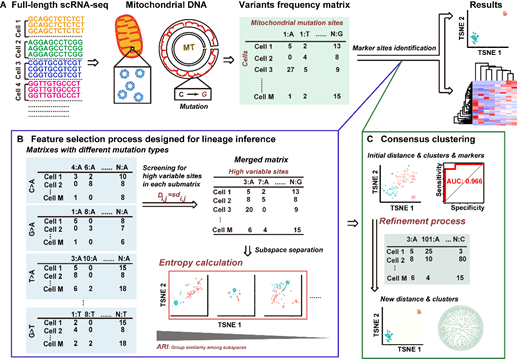
针对上述问题,杨朝勇教授课题组开发了一种以线粒体基因组RNA突变作为内源性条形码的谱系示踪方法(LINEAGE)。LINEAGE采用全长scRNA-seq数据中不同突变类型的高变异位点进行谱系分析,基于突变在细胞上的频率分布模式将其划分到不同的特征子空间。参考信息论中的交叉熵,LINEAGE定义了一种新的‘子空间交叉熵’来量化子空间中分群信息的一致性,并认为交叉熵较低的子空间更可能包含谱系信息。通过一致性聚类整合‘低交叉熵’子空间中的分群信息,从而识别包含谱系信息的标记突变。最后基于标记突变实现细胞亚克隆聚类,获得谱系分析结果。LINEAGE在标准数据集上展示出良好的准确性和高效性,将其应用于一个BRAF V600E突变的黑色素瘤细胞scRNA-seq数据集,发现了GST可能是提高癌细胞对KRAS抑制剂敏感性的新靶点。由于无需辅助数据或构建外源性条形码,LINEAGE相比于已有方法更为简便且应用范围更广,为生物医学研究中重要基因或细胞簇的发现提供了新的思路和工具。
该工作在我院杨朝勇教授、复旦大学生命科学学院李晋教授和上海交通大学分子医学研究院宋佳研究员的共同指导下完成。研究的算法部分由我院2019级博士生林黎完成,GST靶点实验验证由复旦大学张裕丰博士完成。研究工作得到国家重点研发计划项目(2018YFA0801300、2018YFA0801300),国家自然科学基金(32071138、22104080、21735004、21927806)等资助。
论文链接:https://www.pnas.org/content/119/5/e2119767119
